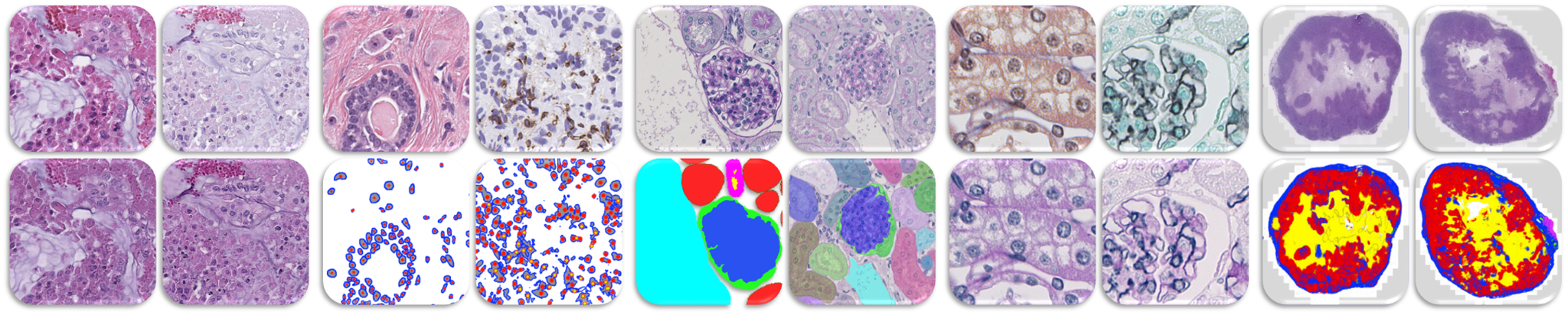
Digitale Pathologie
Motivation
Die großskalige Hochdurchsatz-Digitalisierung von histologischen Glasobjektträgern durch Objektträgerscanner, die digitale Whole Slide Images (WSIs) erzeugen, und verbesserte Software- und Hardware-Lösungen, die zusammen als digitale Pathologie bezeichnet werden, eröffneten neue Möglichkeiten der automatisierten, hochpräzisen und reproduzierbaren Quantifizierung in der Pathologie. Außerdem ermöglicht die digitale Pathologie die wirksame Anwendung von künstlicher Intelligenz (KI), d. h. Computersysteme können Aufgaben ausführen, die normalerweise menschliche Intelligenz erfordern. Maschinelles Lernen und insbesondere Deep Learning (DL) sind Forschungsbereiche der KI mit Schwerpunkt auf Methoden, bei denen sich Computersysteme durch Erfahrung automatisch verbessern. Diese Ansätze werden höchstwahrscheinlich die pathologische Diagnostik in der Zukunft verbessern und transformieren, sodass eine präzise, reproduzierbare und personalisierte Computational Pathology entsteht. Zudem können sie sogar das Potenzial haben, neue diagnostische Merkmale und Biomarker zu identifizieren, die bisher nicht erkannt wurden, wie z. B. in der Onkopathologie.
Wissenschaftliche Fragestellungen
Die Pathologie bleibt eine wichtige Disziplin in der Diagnostik von Krankheiten, während histologische Analysen sowohl bei experimentellen als auch bei klinischen Studien eine wichtige Rolle spielen. Derzeit verlässt sich die Histopathologie noch weitgehend auf die manuelle und oft subjektive Auswertung oder auf herkömmliche computergestützte Verfahren, die nur begrenzte und manchmal ungenaue Ergebnisse liefern. Die modernen DL-Ansätze konzentrieren sich auf überwachte Methoden, die auf dem Training mit großen Datenmengen basieren, die von Experten (Pathologen im Falle der Histologie) beschriftet wurden. Die Sammlung etikettierter medizinischer Daten ist jedoch oft sehr teuer in Bezug auf Zeit und Aufwand der Experten. Unüberwachte Methoden erfordern große Datenmengen, die leider nicht immer verfügbar sind. Außerdem gibt es keine standardisierten Präparations- und Aufnahmeprotokolle für histologische Bilddaten, was zu einer großen Variabilität der Daten zwischen und innerhalb von Laboren führt, wie z. B. Schichtdicke, Färbungsvariabilität, Scanqualität usw. Diese Faktoren begrenzen die Entwicklung von DL-Algorithmen in der Pathologie und Pathomik erheblich.
Abschlussarbeiten
Es werden regelmäßig neue Abschlussarbeiten im Bereich der Digitalen Pathologie ausgeschrieben. Neben der Gesamtübersicht gibt es auch noch zahlreiche, noch nicht ausgeschriebene Themen, die gerne im persönlichen Gespräch vorgestellt werden.
Kooperationspartner
- Prof. Dr. med. Peter Boor, Institut für Pathologie, Universitätsklinikum Aachen
- Dr. rer. nat. Barbara Mara Klinkhammer, Institut für Pathologie, Universitätsklinikum Aachen
- Prof. Dr. med. Friedrich Feuerhake, Institut für Pathologie, Medizinische Hochschule Hannover
- Dr. Julia Schüler, Charles River Discovery Research Services Germany GmbH, Freiburg i. Br.
- Dr. Johannnes Lotz, Fraunhofer-Institut für Digitale Medizin MEVIS, Lübeck
- Dr. Henning Höfener, Fraunhofer-Institut für Digitale Medizin MEVIS, Lübeck
- Dr. André Homeyer, Fraunhofer-Institut für Digitale Medizin MEVIS, Lübeck
Drittmittel
- DFG Klinische Forschungsgruppe, „Integration neuer Methoden zur Verbesserung von translationaler Nierenforschung“, Projektnr. KFO 5011
- BMBF Verbundprojekt: „SynDICAD: Erzeugung Synthetischer Daten für die Anwendung Künstlicher Intelligenz bei Computergestützten Biomarker Analysen in der Digitalen Pathologie“, Projektnr. FKZ: 01IS21067
- BMBF KMU-Innovativ-14 Projekt. „Entwicklung eines innovativen Lungenkarzinom Maus Modell, das die Interaktion des Tumors mit dem Stroma und dem Immunsystem modelliert (ILUMINATE)“ Projektnr. FKZ: 031B0006B
Kontakt
Veröffentlichungen
2022

Nassim Bouteldja, Barbara M. Klinkhammer, Tarek Schlaich, Peter Boor and Dorit Merhof
Improving Unsupervised Stain-To-Stain Translation using Self-Supervision and Meta-Learning
In: Journal of Pathology Informatics
2022

Laxmi Gupta, Barbara Mara Klinkhammer, Claudia Seikrit, Nina Fan, Nassim Bouteldja, Philipp Gräbel, Michael Gadermayr, Peter Boor and Dorit Merhof
Large-scale extraction of interpretable features provides new insights into kidney histopathology – a proof-of-concept study
In: Journal of Pathology Informatics
2021

Barbara M. Klinkhammer Nassim Bouteldja and Dorit Merhof
Deep Learning-Based Segmentation and Quantification in Experimental Kidney Histopathology
In: Journal of the American Society of Nephrology
2020

Daniel Bug, Gregor Nickel, Anne Grote, Friedrich Feuerhake, Eva Oswald, Julia Schüler and Dorit Merhof
Image Quilting for Histological Image Synthesis
In: Bildverarbeitung für die Medizin (BVM)
2020

Daniel Bug, Felix Bartsch, Nadine Sarah Schaadt, Mathias Wien, Friedrich Feuerhake, Julia Schüler, Eva Oswald and Dorit Merhof
Scalable HEVC for Histological Whole-Slide Image Compression
In: Bildverarbeitung für die Medizin (BVM)
2019

Michael Gadermayr, Ann-Kathrin Dombrowski, Barbara Mara Klinkhammer, Peter Boor and Dorit Merhof
CNN Cascades for Segmenting Sparse Objects in Gigapixel Whole Slide Images
In: Computerized Medical Imaging and Graphics 71
2019

Michael Gadermayr, Laxmi Gupta, Vitus Appel, Peter Boor, Barbara Mara Klinkhammer and Dorit Merhof
Generative Adversarial Networks for Facilitating Stain-Independent Supervised & Unsupervised Segmentation: A Study on Kidney Histology
In: IEEE Transactions on Medical Imaging
2019

Michael Gadermayr, Laxmi Gupta, Barbara Mara Klinkhammer, Peter Boor and Dorit Merhof
Unsupervisedly Training GANs for Segmenting Digital Pathology with Automatically Generated Annotations
In: 2nd International Conference on Medical Imaging with Deep Learning (MIDL)
2019

Daniel Bug, Friedrich Feuerhake, Eva Oswald, Julia Schüler and Dorit Merhof
Semi-Automated Analysis of Digital Whole Slides from Humanized Lung-Cancer Xenograft Models for Checkpoint Inhibitor Response Prediction
In: Oncotarget
2019

Daniel Bug, Dennis Eschweiler, Qianyu Liu, Justus Schock, Leon Weninger, Friedrich Feuerhake, Julia Schüler, Johannes Stegmaier and Dorit Merhof
Combined Learning for Similar Tasks with Domain-Switching Networks
In: International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI)
2019

Philipp Gräbel Daniel Bug and Dorit Merhof
Supervised and Unsupervised Cell-Nuclei Detection in Immunohistology
In: 2nd MICCAI Workshop on Computational Pathology (COMPAY)
2019

Laxmi Gupta, Barbara Mara Klinkhammer, Peter Boor, Dorit Merhof and Michael Gadermayr
GAN-Based Image Enrichment in Digital Pathology Boosts Segmentation Accuracy
In: International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI)
2019

Laxmi Gupta, Barbara Mara Klinkhammer, Peter Boor, Dorit Merhof and Michael Gadermayr
Iterative learning to make the most of unlabeled and quickly obtained labeled data in histology
In: 2nd International Conference on Medical Imaging with Deep Learning (MIDL)
2018

Laxmi Gupta, Barbara Mara Klinkhammer, Peter Boor, Dorit Merhof and Michael Gadermayr
Stain Independent Segmentation of Whole Slide Images: A Case Study in Renal Histology
In: IEEE International Symposium on Biomedical Imaging (ISBI)
2018

Michael Gadermayr, Dennis Eschweiler, Barbara Mara Klinkhammer, Peter Boor and Dorit Merhof
Gradual Domain Adaptation for Segmenting Whole Slide Images Showing Pathological Variability
In: International Conference on Image and Signal Processing (ICISP)
2018

Michael Gadermayr, Vitus Appel, Barbara M. Klinkhammer, Peter Boor and Dorit Merhof
Which Way Round? A Study on the Performance of Stain-Translation for Segmenting Arbitrarily Dyed Histological Images
In: International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI)
2017

Michael Gadermayr, Sean Steven Cooper, Barbara Klinkhammer, Peter Boor and Dorit Merhof
A Quantitative Assessment of Image Normalization for Classifying Histopathological Tissue of the Kidney
In: German Conference on Pattern Recognition (GCPR)
2017

Michael Gadermayr, Dennis Eschweiler, Abiramjee Jeevanesan, Barbara Mara Klinkhammer, Peter Boor and Dorit Merhof
Segmenting Renal Whole Slide Images Virtually Without Training Data
In: Computers in Biology and Medicine 90
2017

Daniel Bug, Anne Grote, Julia Schüler, Friedrich Feuerhake and Dorit Merhof
Analyzing Immunohistochemically Stained Whole-Slide Images of Ovarian Carcinoma
In: Bildverarbeitung für die Medizin (BVM)
2017

Daniel Bug, Steffen Schneider, Anne Grote, Eva Oswald, Friedrich Feuerhake, Julia Schüler and Dorit Merhof
Context-based Normalization of Histological Stains using Deep Convolutional Features
In: MICCAI International Workshop on Deep Learning in Medical Image Analysis (DLMIA)
2016

Michael Gadermayr, Barbara M. Klinkhammer, Peter Boor and Dorit Merhof
Do we Need Large Annotated Training Data for Detection Applications in Biomedical Image Data? A Case Study in Renal Glomeruli Detection
In: MICCAI Workshop on Machine Learning in Medical Imaging (MLMI)
2016

Michael Gadermayr, Martin Strauch, Barbara Mara Klinkhammer, Sonja Djudjaj, Peter Boor and Dorit Merhof
Domain Adaptive Classification for Compensating Variability in Histopathological Whole Slide Images
In: International Conference on Image Analysis and Recognition (ICIAR)
2016

Daniel Bug, Julia Schüler, Friedrich Feuerhake and Dorit Merhof
Multi-class single-label classification of histopathological whole-slide images
In: IEEE International Symposium on Biomedical Imaging (ISBI)
2015

Daniel Bug, Friedrich Feuerhake and Dorit Merhof
Foreground Extraction for Histopathological Whole-Slide Imaging
In: Bildverarbeitung für die Medizin (BVM)