Synthese, Segmentierung und Verfolgung von Mikroskopiebildern lebender Zellen
Motivation
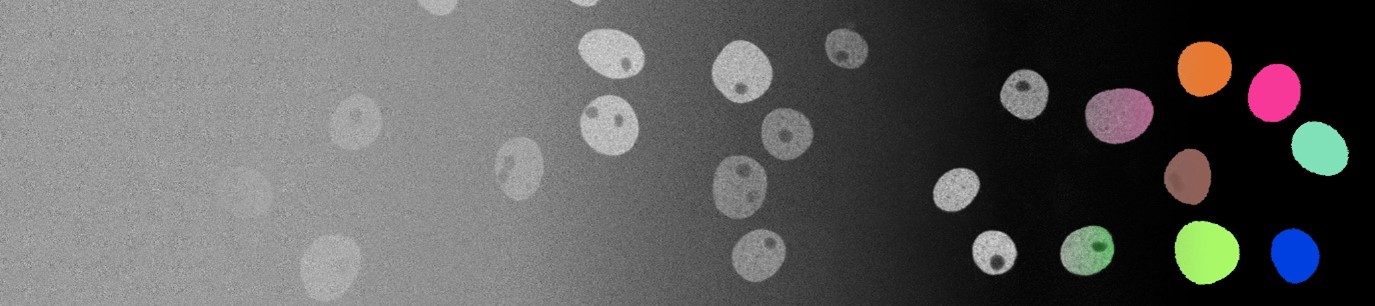
Die Entwicklungsbiologie konzentriert sich darauf, die Prozesse zu untersuchen, wie Zellen migrieren, interagieren und sich teilen. Insbesondere die Analyse von Zellstandort und -morphologie, die durch Segmentierungs- und Verfolgungsansätze analysiert werden, stellt eine der Schlüsselaufgaben beim Verständnis der zugrunde liegenden Prinzipien der Entwicklung dar. Technologische Fortschritte ermöglichen es Biologen, umfangreiche Mengen hochdetaillierter Mikroskopiebilddaten zu erfassen und zu speichern, was die Anwendung automatisierter Ansätze unerlässlich macht. Das Potenzial aktueller auf Deep Learning basierender Ansätze wird jedoch durch die Knappheit vollständig annotierter Bilddatensätze erheblich eingeschränkt. Daher konzentriert sich dieses Projekt auf die Synthese vollständig annotierter Mikroskopievideodatensätze, um den Bedarf an zeitaufwändigen manuellen Annotationsbemühungen zu beseitigen.
Wissenschaftliche Fragestellungen
- Wie lassen sich realistische, vollständig synthetische Zelltexturen und -formen auf der Grundlage einer begrenzten Menge annotierter realer Daten generieren?
- Wie lassen sich die räumlich-zeitlichen Beziehungen von Zellkörpern erfassen und auf den generierten Daten widerspiegeln?
- Wie stellt man sicher, dass im synthetischen Datensatz ausreichend Variabilität vorhanden ist und wie bewertet man dessen Qualität?
- Welche Verbesserungen können erzielt werden, indem Segmentierungs- und Verfolgungsmodelle mit reichlich synthetischen Daten trainiert werden, im Vergleich zu ihrer Schulung mit begrenzten realen Daten?
Kooperationspartner
- G. U. Nienhaus and Dr. A. Kobitski, Institut für Angewandte Physik, Karlsruher Institut für Technologie (KIT)
- Prof. U. Strähle and Dr. M. Takamiya, Institut für Biologische und Chemische Systeme – Biologische Informationsverarbeitung, Karlsruher Institut für Technologie (KIT)
- apl. Prof. R. Mikut, Institut für Automation und Angewandte Informatik, Karlsruher Institut für Technologie (KIT)
Drittmittel
- DFG Sachbeihilfe, “On-the-fly Datensynthese für eine auf Deep Learning basierende Analyse von 3D+t Mikroskopie Experimenten”, Projektnr. 447699143
Kontakt
Veröffentlichungen

Towards Annotation-Free Segmentation of Fluorescently Labeled Cell Membranes in Confocal Microscopy Images
In: MICCAI International Workshop on Simulation and Synthesis in Medical Imaging (SASHIMI)











